关于基于PRS的专业版微解读代码和说明
多基因风险评分分析(Polygenic Risk Score)是一个用来评估个体患某种疾病风险的方法,它是通过综合GWAS统计数据的基因型效应值来计算的。
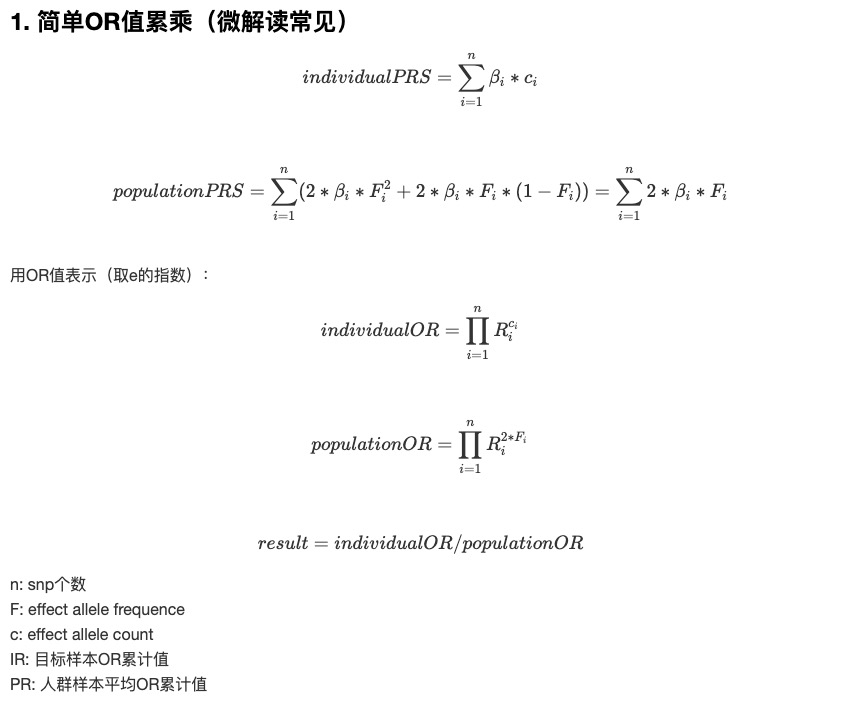
计算公式如下图所示:
做微解读时候应该注意:
1. 我们最终要给出个体风险值相对人群平均风险值的倍数关系,所以最后结果一定是累积OR值相除,而不是累积beta值相除,不然结果会产生负数。
2. 收集GWAS文献中的snp信息时,注意文章给出的effect allele是否是基因组正链的,如果不是需要用碱基互补规则修正过来。
3. 每个位点都需要处理no call或不存在的情况,一般都是用maf值来折算,位点的人群maf可以从公共数据库dbsnp中查得,一般选东亚人群的。
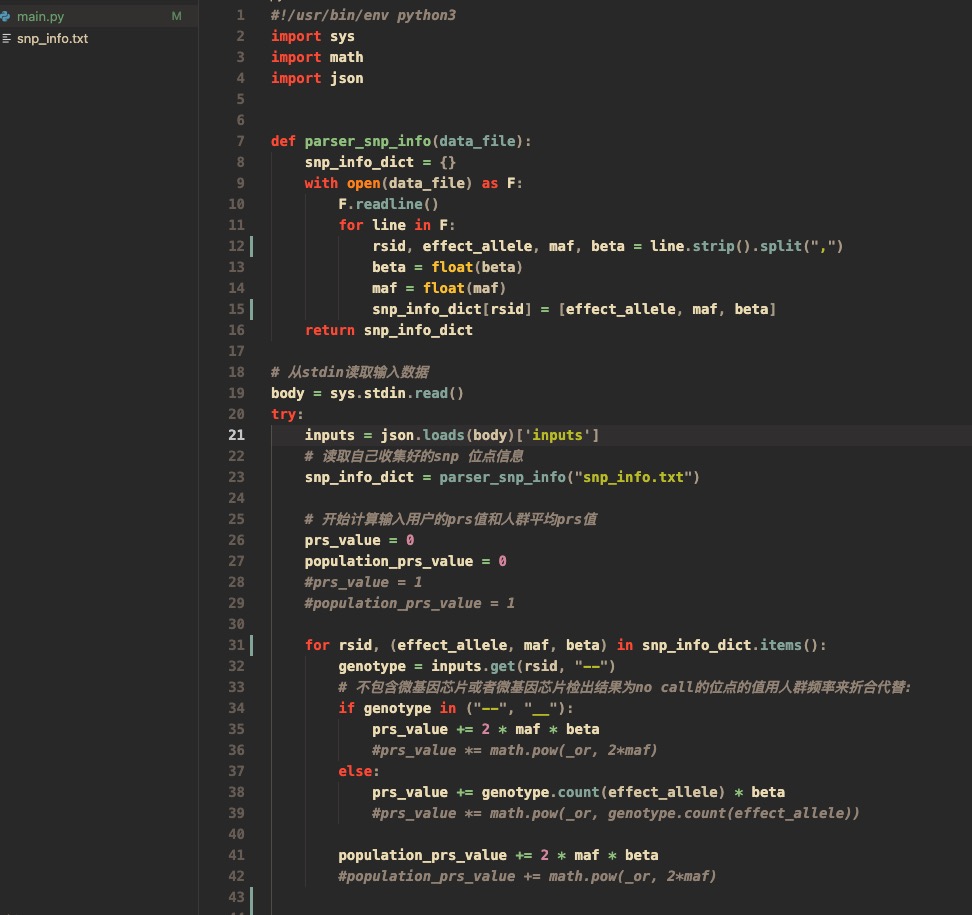
这里给一个示例代码给大家参考下:
代码说明:
1. 正常情况下,下载该源码,只需将snp_info.txt中的snp信息修改成自己收集的信息(格式和列顺序不能改),不做其他更改即可使用。
2. 代码给出的是用beta值来计算,如果GWAS文章给的是OR值, 可先将其转化为beta值(beta = ln(OR)),也可以直接使用OR值计算(示例代码里也有,只是被注释掉了)
3. 另外虽然微解读支持python调用第三方库,但建议只使用python标准库来解决简单的数据处理问题,这样有利于微解读代码审核者发现问题。
代码下载地址:https://github.com/TuBieJun/wegene_weijiedu_example
希望大家都能做出自己的专业版微解读!
计算公式如下图所示:
做微解读时候应该注意:
1. 我们最终要给出个体风险值相对人群平均风险值的倍数关系,所以最后结果一定是累积OR值相除,而不是累积beta值相除,不然结果会产生负数。
2. 收集GWAS文献中的snp信息时,注意文章给出的effect allele是否是基因组正链的,如果不是需要用碱基互补规则修正过来。
3. 每个位点都需要处理no call或不存在的情况,一般都是用maf值来折算,位点的人群maf可以从公共数据库dbsnp中查得,一般选东亚人群的。
这里给一个示例代码给大家参考下:
代码说明:
1. 正常情况下,下载该源码,只需将snp_info.txt中的snp信息修改成自己收集的信息(格式和列顺序不能改),不做其他更改即可使用。
2. 代码给出的是用beta值来计算,如果GWAS文章给的是OR值, 可先将其转化为beta值(beta = ln(OR)),也可以直接使用OR值计算(示例代码里也有,只是被注释掉了)
3. 另外虽然微解读支持python调用第三方库,但建议只使用python标准库来解决简单的数据处理问题,这样有利于微解读代码审核者发现问题。
代码下载地址:https://github.com/TuBieJun/wegene_weijiedu_example
希望大家都能做出自己的专业版微解读!



3 个回复
赞同来自:
赞同来自:
赞同来自:
要回复问题请先登录或注册