【已完结】原始数据的“绽放”——人类疾病各大数据库比对结果
一、概述
拿到wegene原始数据,和人类疾病突变数据库比对,再根进行过滤,最终得到和疾病相关的突变。
二、原料
1、wegene.txt;2、软件、脚本;3、数据库;4、Linux计算机、windows计算机、excel&VBA宏编程等;5、自编写的“基因信息查询系统”(Windows环境下的面向对象可视化);6、手机、耳机和HiFi无损音乐。
三、过程
我的wegene原始数据共有位点596744个,其中有rs号578153个,占约97%,因为绝大部分有rs号,所以按照rs号注释而不按位置,更为方便和不易出错。
第一次过滤(脚本过滤(VBA、perl、awk、grep等))过程:提取rs号进行数据库注释,605096行→删除内含子等、删除同义突变(synonymous SNV),剩下37055行→删掉非突变的位点(野生型纯合),剩下5918行→删掉E东亚人高频率(视为多态)的行,剩下342个位点,对应250个基因。
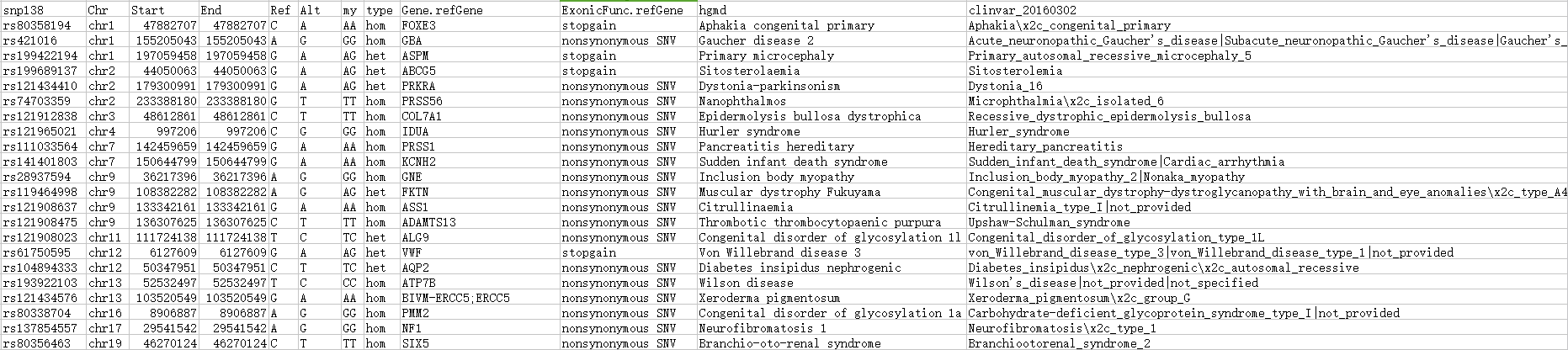
第二次过滤(手动过滤)过程:将基因名(简称)输入自编写的查询系统软件中,可查到数据库注释信息,结合查询信息手动筛出重要的位点。1、删掉未检出位点(--),仅保留外显子、错义突变、stopgain等,余下130个;2、删掉clinvar4个良性突变,余下126个位点,其中杂合子55个,隐性纯合71个;3、仅保留有clinvar信息的行,余下59个位点,其中杂合子24个,对应22个基因,找到2个位点是Rh血型基因(RHD),找到两个OMIM数据库的常染色体隐性遗传但我是杂合子,但是OMIM按基因不按位点,不能确定;4、按clinvar注释的仅保留致病性(Pathogenic)相关位点,余下22行(见图一),其中包括一个可能为携带者但不患病的ABCG5基因,包括几个先天病、婴儿病但我好好地没有任何事儿,其余病均没有临床发现。
图一:过滤结果
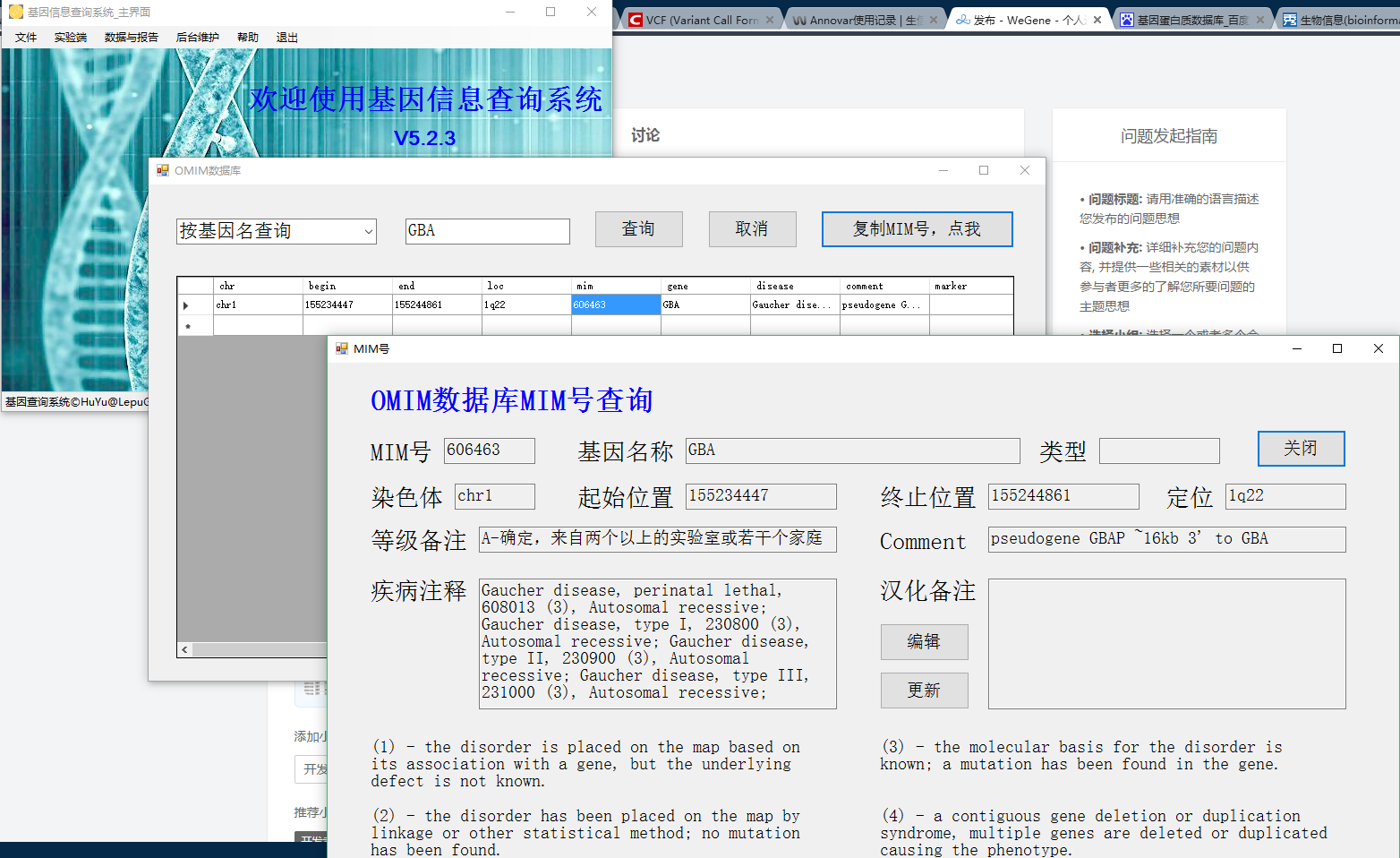
图二:软件示例截图
拿到wegene原始数据,和人类疾病突变数据库比对,再根进行过滤,最终得到和疾病相关的突变。
二、原料
1、wegene.txt;2、软件、脚本;3、数据库;4、Linux计算机、windows计算机、excel&VBA宏编程等;5、自编写的“基因信息查询系统”(Windows环境下的面向对象可视化);6、手机、耳机和HiFi无损音乐。
三、过程
我的wegene原始数据共有位点596744个,其中有rs号578153个,占约97%,因为绝大部分有rs号,所以按照rs号注释而不按位置,更为方便和不易出错。
第一次过滤(脚本过滤(VBA、perl、awk、grep等))过程:提取rs号进行数据库注释,605096行→删除内含子等、删除同义突变(synonymous SNV),剩下37055行→删掉非突变的位点(野生型纯合),剩下5918行→删掉E东亚人高频率(视为多态)的行,剩下342个位点,对应250个基因。
第二次过滤(手动过滤)过程:将基因名(简称)输入自编写的查询系统软件中,可查到数据库注释信息,结合查询信息手动筛出重要的位点。1、删掉未检出位点(--),仅保留外显子、错义突变、stopgain等,余下130个;2、删掉clinvar4个良性突变,余下126个位点,其中杂合子55个,隐性纯合71个;3、仅保留有clinvar信息的行,余下59个位点,其中杂合子24个,对应22个基因,找到2个位点是Rh血型基因(RHD),找到两个OMIM数据库的常染色体隐性遗传但我是杂合子,但是OMIM按基因不按位点,不能确定;4、按clinvar注释的仅保留致病性(Pathogenic)相关位点,余下22行(见图一),其中包括一个可能为携带者但不患病的ABCG5基因,包括几个先天病、婴儿病但我好好地没有任何事儿,其余病均没有临床发现。
图一:过滤结果
图二:软件示例截图


4 个回复
赞同来自:
赞同来自:
赞同来自:
赞同来自:
要回复问题请先登录或注册